一、Primer Premier 5 设计引物
1. 软件或程序
DNAStar/EditSeq、Primer Premier 5、Blast
2. 材料
核酸序列(DNA或cDNA)
3. 操作步骤
(1) 获得核酸序列
进入NCBI→→从GenBank中搜索核酸序列→→选择核酸序列→→复制核酸序列→→粘贴到DNAStar/EditSeq→→保存为Seq文件;
(2) 将核酸序列导入Primer Premier 5
打开Primer Premier 5软件→→显示“Primer Premier”主窗口→→点击“File/New/DNA sequence”→→显示“GeneTank”窗口→→用“Ctrl+C”键从EditSeq中复制核酸序列→→用“Ctrl+V”键粘贴核酸序列到“GeneTank”→→点击“OK”; 或打开Primer Premier 5软件→→显示“Primer Premier”主窗口→→点击“File/Open/DNA sequence”→→显示“Select Multiple DNA Files”窗口→→选定目标序列,点击“Add”→→点击“OK”→→显示“Import Sequence”窗口→→双击任意核酸序列→→显示“GeneTank”窗口;
(3) 自动搜索引物
点击“Primer”→→显示“Primer Premier”窗口→→点击“Search”→→显示“Search Criteria”窗口→→设定相关参数,如Type、Ranges、Length、Mode等→→点击“OK”→→显示“Search Progress”窗口→→点击“OK”→→显示“Search Results”窗口→→根据Rating,察看不同引物对的详细信息→→选择合适引物对;
(4) 人工设计引物
点击“Primer”→→显示“Primer Premier”窗口→→用鼠标点击序列的不同位置→→初步确定引物的位置→→点击“Edit Primers”→→显示“Edit Primer”窗口→→对引物进行修改和分析,如引入酶切位点或碱基突变→→点击“OK”→→显示人工设计引物对;
(5) 在Primer Premier中保存引物
点击主窗口“Function/Save to Database”→→显示“Primer Datebase”窗口→→用“Ctrl”键选定2条引物→→点击“Export”→→选定“Primer Datebase”中1条引物→→点击“Edit Primer”→→显示“Edit Primer”窗口→→输入引物“Name”,如“aHEV1”→→点击“OK”→→同样方法命名另1条引物→→点击“Save Primer Datebase”→→同样方法向“Primer Datebase”中加入和命名其它引物;
(6) 引物序列保存为Word文件
显示“Primer Datebase”→→显示“Edit Primer”窗口→→用“Ctrl+V”键复制引物→→粘贴到Word文件;
(7) 关闭Primer Premier5;
(8) Blast分析
进入NCBI→→Blast分析;
(9) 再次核对引物序列,订购引物。
4. 注意事项
(1) 从GenBank中搜索核酸序列时,尽量选择多个全长的核酸序列,找到保守区,尽量在保守区设计引物。
(2) GenBank中目标核酸序列相关信息,如序列号、作者、来源等,可单独建立一个Word文件,以便于以后查阅。
(3) 引物长度一般18-30bp,GC含量为40-60%,正向、反向引物Tm值越接近越好,最好不要超过5℃。
(4) 引物3’端序列对PCR效率最为重要,不可以引入碱基突变,避免3个及以上的核苷酸重复序列,如GGG、TTTT等。
(5) 引物5’端序列对PCR效率影响较小,可以引入碱基突变,如限制性内切酶位点,这在构建表达载体中常用。
(6) 影响引物质量的四项指标:Hairpin(发卡结构),Dimer(二聚体),False Priming(错配),Cross Dimer(交叉二聚体),引物中出现的指标越少越好,其中“False Priming”最为常见。出现上述指标时,其“△G”绝对值越小越好,通常要求小于10。
(7) 有时软件自动形成的引物并不合适,需要进行人工设计和调整,此时需要耐心分析。
(8) 其它设计引物的软件,如Oligo 6,DNAStar/PrimerSelect,或通过网络获得引物。
(9) 引物的好坏,最终需PCR来证明,对引物要求极高时(如诊断用引物),可同时设计几对引物进行分析比较。
(10)可选择以下公司合成引物:
济南博亚(jinaninvitrogen@163.com,崔全新,13561770809);
上海生工(联系人贾文涛,05386609100)
参考文献
1、J.萨姆布鲁克,D.W.拉塞尔著,黄培堂等译.分子克隆实验指南(第3版)[M].北京:科学出版社,2002.607-609
2、Primer Premier 5 中文使用说明书
整理人:张振杰 审校人:李娟
二、PCR程序
1.仪器耗材
PCR仪、100ul和10ul移液枪及枪头、漩涡振荡器、PCR管等
2.试剂及配制
不同的酶 【如:HS ploymerase(TaKaRa)、r Taq(TaKaRa)】、 5ⅹBuffer(TaKaRa)、d NTP(2.5mmol)(TaKaRa)、引物、DEPC H2O、RT产物等。
3.实验步骤
⑴、引物设计原则:
①长度以15-30个碱基为宜。
②正向反向两条引物链之间的距离适中,一般使扩增出的片断在300-1100bp之间。如果使用RT-PCR检测RNA病毒,引物间距离以500bp最佳。片断过短影响结果判定,过长则不易扩增。
③引物碱基组成应随机分布,G+C含量在45-55%为宜。
④引物自身不形成二级结构,引物间没有互补序列(4个碱基以上)。
⑤引物序列必须是被检病原微生物特异的。
⑥引物3’端碱基与模板DNA一定要配对,并且3’末端碱基最好选T、C或者G,不选A。
⑵、设计程序中用到的温度
变性温度: 94˚C or 95˚C ,不变;
延伸温度: 72˚C,不变;
退火温度: 50˚C左右,可变,且可能变动很大。
4.注意事项
一、PCR出现假阴性原因:
⑴模板因素:①模板中含有杂蛋白;②模板中含有Taq酶抑制剂;③模板中蛋白质残留,特别是染色体中的组蛋白残留;④提取制备模板时丢失过多;⑤模板核酸变性不彻底。
模板因素引起假阴性解决方案:①配制有效而稳定的消化处理液;②提取程序固定,不宜随意改动;③模板DNA的溶解液应固定不变。
⑵酶失活:①酶本身的质量问题(供应商的问题);②酶存放时间太长;③酶存放方式不当,或其他意外原因;④忘记添加Taq酶。
酶失活引起假阴性解决方案:①检查加样程序及过程,看是否忘记加Taq酶;②更换新的Taq酶;③新旧两种Taq酶同时使用。
⑶引物:①引物质量;②引物浓度;③两条引物的浓度是否对称等。
引物引起假阴性解决方案: ①选定一个好的引物合成单位; ②引物浓度不仅要看OD值,稀释时更要平衡其摩尔浓度;③引物应高浓度小量分装保存,防止反复冻融或长期冷冻保存,导致引物的变质降解失效,也可要求合成单位将固体分装;④引物设计不合理,如长度不够,引物本身或两条引物之间形成二聚体等。
⑷Mg2+浓度:Mg2+浓度对PCR扩增效率影响极大,浓度过高可降低PCR扩增的特异性;浓度过低则影响PCR扩增产量甚至使PCR扩增失败,出现假阴性。
Mg2+浓度引起假阴性解决方案:设置一组反应,其中每一反应中的其他离子浓度相同(如Tris-Cl, KCl等),而MgCl2浓度不同(由0.5~6mmol/L,每次增加0.5mmol/L),由此来摸索最佳Mg2+浓度。
⑸反应体积的改变:做小体积PCR后,再做大体积时,一定要重新摸索条件,而非简单地将反应体系中的各个成分加大几倍,否则易失败。
⑹物理原因:①变性温度低,变性时间短;②退火温度过高,影响引物与模板的结合而降低PCR扩增效率;③延伸时间过短等。
物理原因引起假阴性解决方案:①提高变性温度,延长变性时间,特别是首次循环,必要时可设为95℃ ,10min,或PCR反应以前在开水中煮沸几分钟。②退火温度应根据Tm值来设定,也可首先将退火温度设为45℃ ,然后再逐次提高(以2℃ /次为宜)。
③延伸温度以1000bp/min足够,必要时也可适当延长,特别是末次循环,一定要延伸10min。
⑺靶序列变异:靶序列变异导致假引性的情况常常发生在靶序列变异正好发生于特异性引物与之结合部的中间,使引物失效。
解决方案:根据已知序列重新选择区段设计引物。
二、PCR出现假阳性原因:
⑴引物设计不合适:①选择的扩增序列与非目的扩增序列有同源性,从而扩增出非目的性序列的PCR产物。②靶序列太短或引物太短导致特异性不强。
解决方案:选择特异性强的区段设计引物。
⑵靶序列或扩增产物的交叉污染:①整个基因组或大片段的污染;②空气中的小片断核酸污染。
整个基因组或大片段的污染的解决方案:①操作时小心轻柔,防止将靶序列吸入加样枪内或溅出离心管污染环境。②除酶及不耐热物品外,所有试剂及耗材均应高温高压消毒,所用离心管及枪头均应一次性使用。③必要时,加样本前,反应管及试剂用紫外线照射,以破坏存在的核酸。
空气中的小片断核酸污染:这些小片断比靶序列短,但有一定同源性,可互相拼接,与引物互补后,扩增出PCR产物,导致假阳性出现。
解决方案:运用巢式PCR(Nest PCR)来减轻或消除。
三、出现非特异扩增带原因:
⑴ 引物与靶序列不完全互补,或引物聚合形成二聚体;
⑵ Mg2+浓度过高,退火温度过低,PCR循环次数过多;
⑶ 酶的质量不过关,或加Taq酶的量过多。
PCR出现非特异扩增带解决方案:①必要时重新设计合成引物;②减低酶量或调换另一来源的酶;③降低引物量,适当增加模板量,减小循环次数;④适当提高退火温度或采用二温度点法(94 ℃变性,65 ℃左右退火与延伸)。
四、PCR出现片状拖带或涂抹带原因:
⑴酶量过多或酶的质量差;⑵dNTP浓度过高;⑶Mg2+浓度过高,退火温度过低;⑷循环次数过多。
PCR出现片状拖带或涂抹带解决方案①减少酶量或调换另一来源的酶;②减少dNTP浓度;
③适当降低Mg2+浓度;④增加模板量,减少循环次数。
整理人:李娟 审校人:周宏
三、反转录步骤
1.仪器耗材
PCR仪、100ul和10ul移液枪及枪头、漩涡振荡器、PCR管等
2.试剂及配制
AMV、5ⅹ AMV Buffer(TaKaRa)、d NTP(10mmol)(TaKaRa)、RNase inhibition(TaKaRa)、反转录引物、DEPC H2O、总RNA等。
3.实验步骤
根据不同的需要,反转录体系液不同;一般用的体系为20ul;根据AMV所需要的RNA的量进行加样(我们一般都要多加一些,因为所提取的RNA是总的RNA,并不是病毒RNA,所以要多加一些才能保证病毒RNA的量。)以20ul为例,其具体体系如下:
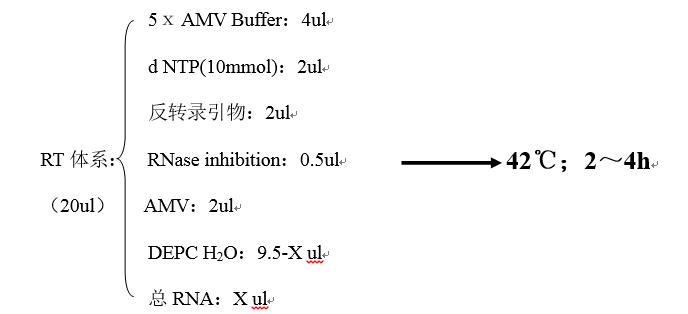
4.注意事项
⑴ 所用的枪头、PCR管和1.5ml的离心管都要用0.1%的DEPC H2O处理,然后高压灭菌;
⑵ 在加样的过程中,要戴口罩和手套,以免口腔和手上的RNA裂解酶降解RNA;
⑶ 其加样顺序为:首先先加DEPC H2O;然后从多到少的顺序加样;最后加总RNA。
整理人:周宏 审校人:张振杰
四、胶回收纯化DNA
1. 仪器耗材
核酸电泳仪、手提式紫外透射仪、天平、离心机、水浴锅、移液枪、手术刀、高压灭菌的1.5ml离心管、枪头。
2. 试剂及配制
胶回收试剂盒、核酸电泳有关试剂。
3. 实验步骤
(1) 样品进行琼脂糖电泳。
(2) 用手提式紫外透射仪照胶,切下含目的条带的胶块,盛于预先称重的1.5ml离心管。
(3) 称量胶块,加适量融胶液在60℃ 左右水浴中融胶,约10min。
(4) 混合胶液加入柱子中,核酸吸附在柱子上。
(5) 离心去掉混合胶液,向柱子中加入适量洗液。
(6) 离心去掉洗液,向柱子中加入适量洗脱液。
(7) 离心得到纯化的核酸。
(8) 琼脂糖核酸电泳鉴定纯化效果。
4.注意事项
(1) 不同试剂盒中试剂存在较大差别,用时注意使用说明。
(2) 样品进行核酸电泳时,使用新鲜的电泳缓冲液,旧的缓冲液会影响纯化效率。
(3) 紫外灯照胶时,选用短波长,时间越短越好。
(4) 切胶时不要切入杂带和多余的胶块。
(5) 加入融胶液后放入水浴锅中,要不时震荡,直至胶块完全融化。
(6) 有的柱子在用前要处理,否则会降低纯化效率。
(7) 每个柱子可吸附约25μg DNA,可多次加入混合胶液,离心后的混合胶液可再次加入柱子中过滤以提高回收率。
(8) 洗脱液用前进行60℃水浴,向柱子中加入时要加到膜中央。
(9) 如果是新试剂盒,注意是否要在洗液中添加无水乙醇。
(10)常用的胶回收试剂盒有:TransGen(全式金)、OMEGA、TAKARA。
整理人:周宏 审校人:张振杰
五、T-A 克隆
1.仪器耗材
恒温摇床,恒温水浴锅,培养平皿90mm,细菌涂布器,冰盒, PCR反应管
2. 试剂及配制
pMD18-T 试剂盒:包括pMD18-T一管 和Solution Ⅰ一管。
PCR产物即target DNA
X-Gal(2%,m/v):
(1)称量1g X-Gal 置于50 ml 离心管中。
(2)加入40ml DMF (二甲基甲酰胺),充分混合溶解后,定容到50ml。
(3)小份分装(1ml/份)后,-20℃避光保存。 用二甲基甲酰胺溶解X-Gal配制成贮存液。IPTG:(0.8mol/L)
8ml蒸馏水溶解2g IPTG,定容至10ml。用0.22m过滤器过滤除菌。
Ampicillin ( 100mg/ml )
(1)称量5g Amplicillin 置于50ml 离心管中。
(2)加入40ml灭菌水充分混合溶解后,定容到50ml。
(3)用0.22μm 过滤膜过滤除菌。
(4)小份分装(1ml/份)后,-20℃保存。
3.实验步骤
(1)在微量离心管中配制下列DNA溶液,全量为5μl
pMD18-T 1μl
Target DNA 1μl
dH2O 3μl
或者target DNA 4μl,而不加dH2O。
(2)加入5μl(等量)的Solution Ⅰ。
(3)16 ℃连接反应60 min或4℃过夜。(若目的条带超过1.5kb,可在16℃条件下适当延长连接反应时间至数小时甚至过夜)
(4)全量(10μl)加入至100μl JM109/DH5a 感受态细胞中冰中放置30min。
(5)42℃加热90秒后,冰中放置2min。
(6)加入890μl SOC/LB培养基,37℃,≦225rpm振荡培养60min。
(7)3000rpm离心2min.弃掉大部分上清,留下大约50-60μl菌液。
(8)菌液涂在含有X-Gal 40μl, IPTG 12ul, Amp的LB-琼脂平板培养基上培养,12-16h形成单菌落。
(9)挑选白色菌落,使用PCR、双酶切、测序等方法确认载体中插入片段的长度大小。
4. 注意事项
在连接反应中
(1)室温(25℃)能正常进行连接反应,但反应效率稍微降低。
(2)5min也能正常进行连接反应,但反应效率稍微降低。
(3)solution I 使用时请与冰中溶解。
(4)插入片段如果挺大,可以考虑适当增加连接时间。
(5)有时可以考虑不用X-Gal,和IPTG,直接挑取单菌落进行PCR筛选。
(6)热激是一个关键步骤,准确地达到热激温度非常重要。此期间绝对不要摇动管。
(7)X-Gal 40ul, IPTG 12ul 菌液涂板之前半小时涂在含有Amp.的板上。
参考文献:
[1]《分子克隆》(第三版)98-99。
[2]《TaKaRa商品目录》中pMD18-T kit使用说明书,L-4—L-5。
整理人:张振杰 审校人:李娟
